Las siglas CRISPR/Cas9 provienen de Clustered Regularly Interspaced Short Palindromic Repeats, en español “Repeticiones Palindrómicas Cortas Agrupadas y Regularmente interespaciadas.” La segunda es el nombre de una serie de proteínas, principalmente unas nucleasas, que las llamaron así por CRISPR associated system (es decir: “sistema asociado a CRISPR”).
¿Cómo surgió?
Todo comenzó en 1987 cuando se publicó un artículo en el cual se describía cómo algunas bacterias (Streptococcus pyogenes) se defendían de las infecciones víricas. Estas bacterias tienen unas enzimas que son capaces de distinguir entre el material genético de la bacteria y el del virus y, una vez hecha la distinción, destruyen al material genético del virus.
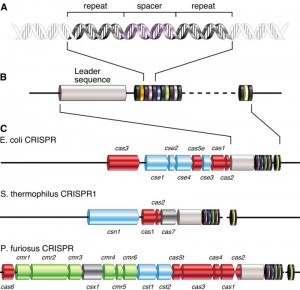
Sin embargo, las bases de este mecanismo no se conocieron hasta más adelante, cuando se mapearon los genomas de algunas bacterias y otros microorganismos. Se encontró que una zona determinada del genoma de muchos microorganismos, sobre todo arqueas, estaba llena de repeticiones palindrómicas(que se leen igual al derecho y al revés) sin ninguna función aparente. Estas repeticiones estaban separadas entre sí mediante unas secuencias denominadas “espaciadores” que se parecían a otras de virus y plásmidos. Justo delante de esas repeticiones y “espaciadores” hay una secuencia llamada “líder”. Estas secuencias son las que se llamaron CRISPR (“Repeticiones Palindrómicas Cortas Agrupadas y Regularmente interespaciadas”). Muy cerca de este agrupamiento se podían encontrar unos genes que codificaban para un tipo de nucleasas: los genes cas.
Cuando un virus entra dentro de la bacteria toma el control de la maquinaria celular y para ello interacciona con distintos componentes celulares. Pero las bacterias que tienen este sistema de defensa tienen un complejo formado por una proteína Cas unida al ARN producido a partir de las secuencias CRISPR. Entonces el material génico del virus puede interaccionar con este complejo. Si ocurre eso, el material genético viral es inactivado y posteriormente degradado. Pero el sistema va más allá. Las proteínas Cas son capaces de coger una pequeña parte del ADN viral, modificarlo e integrarlo dentro del conjunto de secuencias CRISPR. De esa forma, si esa bacteria (o su descendencia) se encuentra con ese mismo virus, ahora inactivará de forma mucho más eficiente al material genético viral. Es, por lo tanto, un verdadero sistema inmune de bacterias.
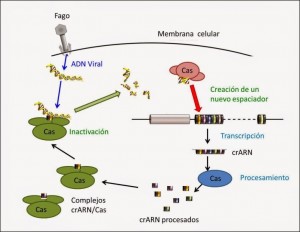
Proceso por el que el sistema CRISPR/Cas9 inactiva virus e integra parte de sus secuencias en el genoma de la bacteria.
Durante los años subsiguientes se continuó la investigación sobre este sistema, pero no fue hasta el año 2012 en el que se dio el paso clave para convertir este descubrimiento, esta observación biológica en una herramienta molecular útil en el laboratorio. En agosto de este año un equipo de investigadores dirigido por las doctoras Emmanuelle Charpentier en la Universidad de Umeå y Jennifer Doudna, en la Universidad de California en Berkeley, publicó un artículo en la revista Science el que se demostraba cómo convertir esa maquinaria natural en una herramienta de edición “programable”, que servía para cortar cualquier cadena de ADN in vitro. Es decir, lograban programar el sistema para que se dirigiera a una posición específica de un ADN cualquiera (no solo vírico) y lo cortaran.
La manera en que lo lograron es demasiado compleja para lo que pretende este blog. Baste simplemente decir que se utilizan unos ARNs que dirigen el sistema hacia el ADN que hay que cortar.
¿Cómo se edita el ADN con esta tecnología?
Todo comienza con el diseño de una molécula de ARN (CRISPR o ARN guía) que luego va a ser insertada en una célula. Una vez dentro reconoce el sitio exacto del genoma donde la enzima Cas9 deberá cortar.
El proceso de editar un genoma con CRISPR/Cas9 incluye dos etapas. En la primer atapa el ARN guía se asocia con la enzima Cas9. Este ARN guía es específico de una secuencia concreta del ADN, de tal manera que por las reglas de complementariedad de nucleótidos se hibridará en esa secuencia (la que nos interesa editar o corregir). Entonces actúa Cas9, que es una enzima endonucleasa (es decir, una proteína que es capaz de romper un enlace en la cadena de los ácidos nucléicos), cortando el ADN. Básicamente podemos decir que el ARN guía actúa de perro lazarillo llevando a Cas9, el ejecutor, al sitio donde ha de realizar su función.
En la segunda etapa se activan al menos dos mecanismos naturales de reparación del ADN cortado. El primero llamado indel (inserción-deleción) hace que, después del sitio de corte (la secuencia específica del ADN donde se unió el ARN guía), bien aparezca un hueco en la cadena, bien se inserte un trocito más de cadena. Esto conlleva a la perdida de la función original del segmento de ADN cortado.
Un segundo mecanismo permite la incorporación de una secuencia concreta exactamente en el sitio original de corte. Para esto, lógicamente, hemos de darle a la célula la secuencia que queremos que se integre en el ADN.
Problemas que puede tener
En principio tiene dos pegas técnicas que han de ser corregidas (ya se está en ello). La primera viene derivada del hecho de que la especificidad del ARN guía no es total. Es decir, este ARN, puede hibridar, juntarse con más de un sitio en el genoma, lo que llevaría a que la enzima Cas9 cortara en un sitio que no nos interese. Hay que recordar que el genoma está compuesto por sucesiones de cuatro “letras”, A, T, G, y C, y la posibilidad de que se repita determinada secuencia es alta, sobre todo si la secuencia no es muy larga (es probable que, por ejemplo, la secuencia AAATGGCAATC esté en más de un gen. La probabilidad de repetición disminuye si aumento la longitud de la “palabra”). El segundo punto débil de la técnica se debe a que Cas9 pueda cortar sin que esté presente el ARN guía. Esto se soluciona con enzimas más precisas.
¿Para qué vale?
De manera molecular podemos decir que esta herramienta se podrá utilizar para regular la expresión génica, etiquetar sitios específicos del genoma en células vivas, identificar y modificar funciones de genes y corregir genes defectuosos. También se está ya utilizando para crear modelos de animales para estudiar enfermedades complejas como la esquizofrenia, para las que antes no existían modelos animales.
¿Cómo nos va a cambiar la vida?
Las posibilidades son casi inimaginables. Con la tecnología CRISPR/Cas9 se inaugura una nueva era de ingeniería genética en la que se puede editar, corregir, alterar, el genoma de cualquier célula de una manera fácil, rápida, barata y, sobre todo, altamente precisa. Cambiar el genoma significa cambiar lo esencial de un ser, recordadlo.
En un futuro relativamente cercano servirá para curar enfermedades cuya causa genética se conozca y que hasta ahora eran incurables. Es lo que seguramente muchas veces habréis oído nombrar como terapia génica. Ya se está trabajando con esta tecnología en estas enfermedades como la Corea de Huntington, el Síndrome de Down o la anemia falciforme. Otra aplicación aparentemente futurista, pero no tan quimérica es la reprogramación de nuestras células para que corten el genoma del VIH.
Así, el MIT (Instituto Tecnológico de Massachussets) anunció ya en marzo de 2014 que había conseguido curar a un ratón adulto de una enfermedad hepática (tirosinemia de tipo I) de origen genético utilizando esta tecnología.
Y, sí, como muchos estáis pensando, esta técnica también vale para modificar los genomas de embriones humanos. Sobre las implicaciones éticas y sociales habría que escribir muchos libros, pero no es nuestro objeto aquí.
Además de estas implicaciones sanitarias, también se puede utilizar para mejorar los alimentos transgénicos (desarrollar nuevas variedades de plantas y animales con características genéticas concretas), modificar bacterias y otros microorganismos de uso industrial y alimentario.
Guerra de patentes
La ciencia también tiene una parte “fea”. Y en esta ocasión, como casi siempre, viene derivada de los intereses económicos (obvios) que tiene la tecnología CRISPR/Cas9.
Poco después del famoso artículo de Doudna y Charpentier, en enero de 2013 los laboratorios de George Church en Harvard y Feng Zhang en el Broad Institute del MIT fueron los primeros en publicar artículos demostrando que CRISPR/Cas9 servía para células humanas. Doudna publicó lo propio de manera independiente apenas unas semanas más tarde.
En abril de 2014 Zhang y el Instituto Broad obtuvieron la primera de entre varias patentes generales que cubren el uso de CRISPR en eucariotas. Eso les otorgaba los derechos para usar CRISPR en ratones, cerdos, humanos… prácticamente en cualquier criatura que no fuera una bacteria.
La velocidad de obtención de la patente sorprendió a algunos. Y fue porque el Instituto Broad había pagado de una manera discreta para que la revisaran muy rápido, en menos de seis meses. Además el proceso se llevó a cabo de una manera casi “secreta”. Junto con la patente llegaron más de mil páginas de documentos. Doudna había presentado antes que la de Zhang, una solicitud de patente. Pero, según Zhang, la predicción de Doudna en su solicitud de que su descubrimiento funcionaría en humanos era una “mera conjetura” y que en cambio él fue el primero en demostrarlo en un acto de invención distinto y “sorprendente”. Para demostrar que fue “el primero en inventar” el uso de CRISPR-Cas en células humanas, Zhang presentó fotos de cuadernos de laboratorio que según él demuestran que tenía el sistema en marcha a principios de 2012, incluso antes de que Doudna y Charpentier publicaran sus resultados o solicitaran su propia patente. Esa cronología significaría que él descubrió el sistema CRISPR-Cas independientemente. En una entrevista, Zhang afirmó que había hecho los descubrimientos él solo. Al preguntársele qué había aprendido del artículo de Doudna y Charpentier, dijo “no mucho”.
Por otra parte, los abogados de Doudna y Charpentier no se van a quedar de brazos cruzados y se espera que monten un “procedimiento de interferencia” en Estados Unidos, que es un proceso legal en el que el ganador se lo lleva todo y en el que un inventor puede hacerse con la patente de otro.
En el juego de las patentes aparecen implicadas tres start-up para las que el control de esas patentes es clave. Entre esas empresas están Editas Medicine, Intellia Therapeutics, ambas de Cambridge, y CRISPR Therapeutics, una start-up de Basilea (Suiza) cofundada por Charpentier. Zhang cofundó Editas Medicine, que en diciembre de 2014 anunció que había comprado la licencia para el uso de su patente al Instituto Broad. Pero Editas no tiene el monopolio sobre CRISPR porque Doudna también fue cofundadora de la empresa. Y desde que salió la patente de Zhang, Doudna ha roto con la empresa, y su propiedad intelectual en forma de su propia patente pendiente de aprobación se ha vendido bajo licencia a Intellia. Para complicar aún más las cosas, Charpentier vendió sus derechos de la misma solicitud de patente a CRISPR Therapeutics.
Por otra parte, cada vez hay más voces que piden que, debido a la gran capacidad de curar enfermedades por parte de CRISPR-Cas9, la tecnología no quede protegida por patente y se deje como acceso público. La propia Charpentier asegura que la tecnología ha sido puesta libremente a disposición de la comunidad investigadora, por lo que no cree que la patente suponga ningún obstáculo al avance científico.
Veremos qué nos depara el futuro.
FUENTE: http://dciencia.es/que-es-la-tecnologia-crispr-cas9/